
Neue prognostische Marker und therapeutische Angriffsziele beim Prostatakarzinom?
Mit der Entdeckung rekurrenter Genfusionen eines Androgen-regulierten 5´-Gens mit einem Gen aus der ETS-Familie beim Prostatakarzinom, scheint ein bedeutender Mechanismus für die Überexpression von onkogen wirkenden ETS-Tanskriptionsfaktoren gefunden zu sein. Hiervon werden bedeutende neue Einsichten in die Biologie dieser Tumorentität sowie Einflüsse auf den klinischen Umgang mit der Krankheit erwartet. Eine Reihe von Daten zeigt, dass durch Fusionsgene offenbar bestimmte molekulare Subtypen charakterisiert werden. Ferner mehren sich Indizien dafür, dass Genfusionen beim Prostatakarzinom einen charakteristischen klinischen Verlauf definieren, und ihr Nachweis somit als diagnostischer Test und Biomarker mit prognostischer Relevanz eingesetzt werden kann. Zudem besteht die Hoffnung, Genfusionen könnten sich für zielgerichtete Therapien als nützlich erweisen.
-
Das gestiegene Bewusstsein der Männer für Prostatakrebs in Verbindung mit der zunehmenden Anwendung des Prostata-spezifischen Antigen (PSA)-Tests
hat dazu geführt, dass Prostatakrebs heute immer häufiger in einem frühen Stadium entdeckt wird. Unentdeckt verliefen solche Tumoren zu einem beträchtlichen
Anteil nicht letal. Andererseits ist die Mortalitätsrate beim Prostatakarzinom noch immer sehr hoch. Prostatakrebs ist die zweithäufigste tumorbedingte
Todesursache beim Mann. Um Überbehandlungen zu vermeiden, aggressive Tumoren aber adäquat zu behandeln, fehlt es bislang an Möglichkeiten, verlässliche
Aussagen über den Verlauf der Krankheit im Einzelfall machen zu können. Die Forschung sucht daher unablässig nach klinisch relevanten Biomarkern, anhand
derer der Verlauf einer Prostatakrebserkrankung vorhersehbar ist.
Genfusionen sind ein häufiges Ereignis bei hämatologischen (Philadelphia-Chromosom) und mesenchymalen malignen Krankheiten. Ihr Vorkommen in Karzinomen war
bis vor kurzem hingegen weitgehend unbekannt. Das hat sich seit ca. drei Jahren mit der Entdeckung zahlreicher rekurrenter genomischer Rearrangements in
Prostatakarzinomen grundlegend geändert. An diesen Umlagerungen sind jeweils ETS-Gene beteiligt. Diese Gene kodieren für eine Gruppe von onkogen wirkenden
Transkriptionsfaktoren. Als Partner in Genfusionen wurde hiervon zunächst entweder ERG (Estrogen-reguliertes Gen), ETV1 oder ETV4
identifiziert [Reviews 1-3].
-
Erst vor drei Jahren erschien der erste Bericht über rekurrente genomische Rearrangements beim Prostatakarzinom, die zur Fusion des 5’-nicht-translatierten
Endes von TMPRRS2 mit einem Gen aus der ETS-Familie führen [4]. Ihre Entdeckung gelang mithilfe des COPA (Cancer Outlier Profile Analysis)-Algorithmus. Die
Methode wurde entwickelt, um Gene zu identifizieren, die nur in einer Untergruppe von Karzinomen in hohem Maße exprimiert werden.
Das TMPRSS2-Gen kodiert für eine Serin-Protease und wird prostataspezifisch stark Androgen-abhängig exprimiert. Eine niedrige intraprostatische Konzentration an Dihydrotestosteron (DHT), wie sie unter einer Androgendeprivationstherapie vorliegt, reicht aus, um eine ETS-Überexpression zu verursachen.
Häufigster Fusionspartner für TMPRSS2 aus der Gruppe der ETS-Gene ist ERG. Beide Gene sind auf dem langen Arm von Chromosom 21 (21q22.2-3) lokalisiert. Als häufigster Fusionsmechanismus gilt die Deletion des kurzen Abschnitts zwischen den beiden Genen.
TMPRSS2:ERG-Fusionen kommen bei ca. jedem zweiten Prostatakarzinom vor. Dieser hohe Anteil ergibt sich aus den gemittelten Ergebnissen von mehr als zwei Dutzend Studien mit insgesamt etwa 1.500 analysierten Fällen von lokalisiertem Prostatakrebs. Er kann je nach untersuchtem Krankengut in weiten Grenzen variieren. Angesichts der hohen Prävalenz von Prostatakrebs ist die TMPRSS2:ERG-Genfusion aber in jedem Fall eine der häufigsten somatischen, genetischen Alterationen im Zusammenhang mit malignen Krankheiten.
Die TMPRSS2:ERG-Fusion kommt häufig in Verbindung mit morphologischen Merkmalen eines aggressiven Prostatakarzinoms vor. Hierzu zählen bläulich getönter Schleim, siebförmige Wachstumsmuster, Makronukleoli, intraduktale Tumorausbreitung sowie Charakeristika von Siegelringzellen. Nur 24% der Tumoren, die keines dieser Merkmale aufweisen, enthalten die TMPRSS2:ERG-Fusion [5].
Rajput et al. (2007) entdeckten TMPRSS2:ERG-Fusionen vorwiegend in mäßig bis schlecht differenzierten Tumoren. Keine Genfusionen fanden sich dagegen
in hyperplastischen Prostatae [6].
-
Die TMPRSS2:ETS-Genfusion kann im Prostatagewebe, in zirkulierenden Prostatakrebszellen und in Urinproben nach Prostatamassage nachgewiesen werden. Für den Nachweis
eignen sich zwei Techniken, die auf unterschiedlichen Ebenen agieren:
• Auf genomischer Ebene dient die Fluoreszenz-in-situ-Hybridisierung (FISH)-zur Entdeckung chromosomaler Veränderungen.
• Auf Transkriptomebene dienen Polymerase-Kettenreaktion (PCR)-basierte Assays zur Entdeckung und Quantifizierung von Fusionstranskripten.
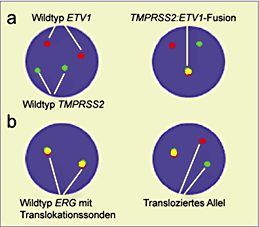
Die Gene für ETV1 und ETV4 liegen jeweils auf einem anderen Chromosom als TMPRSS2. Die FISH-Signale für jedes Gen beider Allele liegen im Zellkern räumlich getrennt voneinander. Durch unterschiedlich farbliche (grün, rot) Markierung von TMPRSS2 und ETV1 bzw. ETV4 lassen sich die Genpaare in Zellen ohne Genfusion (Wildtyp) deutlich unterscheiden. Beim Auftreten eines Signals aus zwei eng beieinanderliegenden Markierungen (grün und rot), dessen Überschneidung gelb erscheint, kann auf eine heterozygote Fusion geschlossen werden (Abb. 1a).
Um mit FISH eine TMPRSS2:ERG-Genfusion entdecken zu können, werden Translokationsassays (break-apart assays) angewandt. Denn die beiden Gene liegen auf Chromosom 21 zu
eng beieinander, als dass die Fusion mittels eines Fusionsassays einwandfrei unterschieden werden könnte. Aus einer Translokation wird in diesem Fall auf eine Fusion
zwischen TMPRSS2 und ERG geschlossen (Abb. 1b).
|
Abb. 1a, b: Assays zur Detektion von Fusionsgenen zwischen TMPRSS2 und ETS-Genen mittels Fluoreszenz-in-situ-Hybridisierung. a) Beispiel für ETV1 bzw. ETV4. b) Beispiel für ERG. |
-
Tomlins et al. (2007) berichteten über weitere 5’-Fusionspartner von ETV1: SLC45A3, HERV-K_22q11.23, C15ORF21, HNRPA2B1 [7].
Diese sind auf unterschiedliche Weise Androgen-reguliert (Androgen-induziert, Androgen-supprimiert, Androgen-insensitiv).
Zunächst waren nur Fusionsgene mit ETV1 und den neuen 5’-Partnern bekannt. Jüngst wurde auch ETV5 als Fusionspartner von TMPRSS2 und SLC45A3 identifiziert. Damit ist neben ETV1 und ETV4 das dritte und letzte Mitglied aus der PEA3-Unterfamilie von ETS-Genen an Genfusionen in Prostatakarzinomen beteiligt [8].
Bislang waren stets nur Transkripte der Fusionsgene identifiziert worden. Mit DDX5-ETV4 wurde erstmals ein Gesamtfusionsprotein identifiziert [9].
-
Genfusionen kommen in den meisten Prostatakarzinomen vor, so dass mit ihrer Entdeckung sogleich über ihre mögliche Rolle bei der Tumorigenese spekuliert wurde.
Es wird hypothetisiert, dass die 5’-nicht-translatierte Region von TMPRSS2, die die Elemente für die prostataspezifische Expression der kodierten Serin-Protease
enthält, in TMPRSS2:ETS-positiven Prostatakrebszellen die Überexpression von ETS-Transkriptionsfaktoren vorantreibt.
Perner et al. (2007) analysierten Gewebeproben von Patienten mit benigner Prostataerkrankung, Prostatakrebs-Vorstufen, klinisch lokalisierten Prostatakarzinomen,
sowie Hormon-naive und Hormon-refraktäre Metastasen. In ca. jedem zweiten lokalisiertem Prostatakarzinom (48,5%) sowie in 30% der Hormon-naiven und 33% der
Hormon-refraktären Metastasen wurde die TMPRSS2:ERG-Fusion nachgewiesen. Keine Genfusionen wurden in den Gewebeproben bei benigner Prostataerkrankung vorgefunden.
Aber in 19% der prostatischen intraepithelialen Neoplasien (PIN) mit hohem Tumorgrad waren TMPRSS2-ERG-Fusionen nachweisbar (allerdings nur vermischt mit Krebsherden,
die das gleiche ERG-Rearrangement aufwiesen). Offenbar führen genetische Schäden zu deregulierter Proliferation und dem Erscheinungsbild von PIN, während
TMPRSS2:ERG-Fusionen ein frühes Ereignis in der Entwicklung eines invasiven Adenokarzinoms der Prostata sind [10].
-
In einer Reihe von Studien wurde bereits untersucht, ob bzw. inwieweit sich TMPRSS2:ERG eignet, Aussagen über den Krankheitsverlauf machen zu können. Die Ergebnisse
sind nicht frei von Widersprüchen. Doch mehrheitlich wurde das Vorkommen des Fusionsgens mit einer schlechteren Prognose in Zusammenhang gebracht als das Fehlen des Fusionsgens.
Nam et al. (2007) untersuchten bei 26 Patienten mit einem klinisch lokalisierten Prostatakarzinom gleichen histologischen Grades (Gleason Score 7) das Auftreten eines biochemischen Rezidivs nach radikaler Prostatektomie. In elf der Fälle (42,3%) wurde das TMPRSS2:ERG-Fusionsgen nachgewiesen. Bei diesen Patienten war die 5-Jahres-Rezidivrate signifikant höher als bei den Patienten ohne die Genfusion (79,5 versus 37,5%) [11].
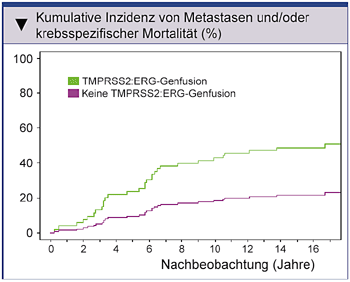
Ergebnisse von Demichelis et al. (2007) lassen erkennen, dass die TMPRSS2:ERG-Fusion beim Prostatakarzinom zur Entwicklung eines aggressiven Phänotyps beiträgt.
Zugleich wird die kritische Rolle von ERG als ein Onkogen bei Prostatakrebs beleuchtet. In einer Kohorte von Prostatakrebs-Patienten unter aktiver Überwachung
(Watchful Waiting) war die kumulative Inzidenz von Metastasierung und/oder krebsspezifischer Mortalität in Fällen mit nachgewiesener TMPRSS2:ERG-Fusion signifikant
höher (Abb. 2) [12].
|
Abb. 2: TMPRSS2:ERG ist bestimmender Faktor für die kumulative Inzidenz der Metastasierung und Prostatakrebs-spezifischen Mortalität (nach Demichelis F, et al. 2007). |
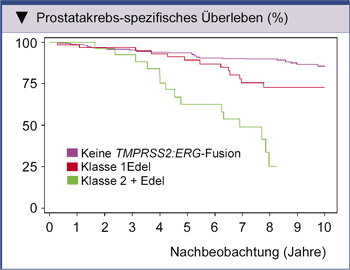
Attard et al. (2008) identifizierten eine neue Kategorie von Prostatakarzinomen, die sich durch eine Duplikation der Fusion von TMPRSS2 mit ERG-Sequenzen in Verbindung mit interstitiellen Deletionen von Sequenzen 5’ von ERG auszeichnet. Diese als 2 + Edel bezeichnete ERG-Modifikation wird durch zwei oder mehr solitäre 3’-ERG-Kopien pro Zelle in FISH-Analysen definiert. Ihr Auftreten bei 6,6% der untersuchten Fälle war mit einer wesentlich ungünstigeren Prognose behaftet als bei Vorliegen nur einer 3’-ERG-Kopie (1Edel) pro Zelle (Abb. 3) [13].
|
Abb. 3: Kaplan-Meier-Analyse zum Vergleich des Prostatakrebs-assoziierten Überlebens bei Patienten mit einem Prostatakarzinom ohne TMPRSS2:ERG-Fusion sowie mit einer 1Edel- oder einer 2 + Edel-Mutation (siehe Erklärung im Text) (nach Attard G, et al. 2007). |
Es existieren bereits Tests, um Genfusionen in Prostatabiopsien oder Urinproben nachweisen zu können. Inwieweit ihrer Entdeckung prognostische Bedeutung zukommt, muss noch in umfangreichen weiteren Untersuchungen erforscht werden.
Literatur:
[1] Kumar-Sinha C, Tomlins SA, Chinnaiyan AM, 2008. Recurrent gene fusion in prostate cancer. Nature Reviews Cancer 8:497-511.
[2] Morris DS, Tomlins SA, Montie JE, Chinnaiyan AM, 2008. The discovery and appli-ction of gene fusion in prostate cancer. BJU Int 102:276-282.
[3] Perner S, Schmidt FH, Hofer MD, et al. 2007. Die TMPRSS2-ETS-Genfusion beim Prostatakarzinom. Urologe 46:754-760.
[4] Tomlins SA, Rhodes DR, Perner S, et al. 2005. Recurrent fusion of TMPRSS2 and ETS transcription factor genes in prostate cancer. Science 310:644-648.
[5] Mosquera J-M, Perner S, Demichelis F, et al. 2007. Morphological features of TMPRSS2-ERG gene fusion prostate cancer. J Pathol 212:91-101.
[6] Rajput AB, Miller MA, De Luca A, et al. 2007. Frequency of the TMPRSS2:ERG gene fusion is increased in moderate to poorly diffe-rentiated prostate cancers. J Clin Pathol 60:1238-1243.
[7] Tomlins SA, Laxman B, Dhanasekaran SM, et al. (2007). Distinct classes of chromo-somal rearrangements create oncogenic ETS gene fusions in prostate cancer. Nature 448:595-599.
[8] Helgeson BE, Tomlins SA, Shah N, et al. 2008. Characterization of TMPRSS2:ETV5 and SLC45A3:ETV5 gene fusion in prostate cancer. Cancer Res 68:73-80.
[9] Han B, Mehra R, Dhanasekaran SM, et al. 2008. A fluorescence in situ Hybridiza-tion screen foe E26 transformation-specific aberrations: identification of DDX5-ETV4 fu-sion protein in prostate cancer. Cancer Res 68:7629-7637.
[10] Perner S, Mosquera J-M, Demichelis F, et al. 2007. TMPRSS2-ERG fusion prostate cancer: an early molecular event associated with invasion. Am J Surg Pathol 31:882-888.
[11] Nam RK, Sugar L, Wang Z, et al. 2007. Expression of TMPRSS2:ERG gene fusion in prostate cancer cells is an important prognostic factor for cancer progression. Cancer Biol Ther 6:40-45.
[12] Demichelis F, Fall K, Perner S, et al. 2007. TMPRSS2:ERG gene fusion associated with lethal prostate cancer in a watchful waiting cohort. Oncogene 26:4596-4599.
[13] Attard G, Clark J, Ambroisine L, et al. 2008. Duplication of TMPRSS2 to ERG sequen-ces identifies fatal human prostate cancer. On-cogene 27:253-263.
© 2003-2025 pro-anima medizin medien
–
impressum
–
mediadaten
–
konzeption
–
datenschutz
