
Entwicklung und Validierung genomischer Klassifizierer
Anhand archivierter Gewebeproben von Männern, die sich an der Mayo Clinic einer radikalen Prostatektomie aufgrund eines lokalisierten Prostatakarzinoms unterzogen hatten, wurde ein genomischer Klassifizierer mit 22 Markern entwickelt, der auch zahlreiche, nicht kodierende RNA-Sequenzen enthält. In mehreren Studien wurde dessen überlegener prognostischer Nutzen gegenüber herkömmlichen pathologischen Variablen validiert. Der unmittelbar postoperativ anwendbare Test kann anhand des Primärtumors frühzeitig Patienten selektieren, die von intensiveren Therapien profitieren können.
Bei Prostatakrebs-Patienten, die sich der radikalen Prostatektomie unterziehen, entscheidet sich nach der
Operation die Klassifizierung ihrer Krankheit als mit hohem oder niedrigem klinischem Risiko behaftet. Hierzu
werden gewöhnlich pathologische Merkmale wie der Gleason Score 7, extrakapsuläre Ausbreitung, positive Schnittränder,
Invasion der Samenblasen und positiver Nodalstatus herangezogen, die auf ein erhöhtes Risiko der klinischen
Metastasierung hindeuten. Andererseits sind klinisch-pathologische Merkmale und auch ein biochemisches Rezidiv
beim Prostatakrebs trotz hoher Sensitivität keine spezifischen Prädiktoren für Metastasierung und Letalität.
Deshalb wird nach verbesserten Möglichkeiten der Klassifizierung von Prostatakrebs nach radikaler Prostatektomie
geforscht. Insbesondere werden hierbei Veränderungen im Genom der Krebszellen im Zusammenhang mit
Genexpressions-
-
Bei einem Großteil der Prostatakrebs-Patienten finden sich nach der radikalen Prostatektomie ungünstige pathologische
Merkmale. Das bedingt zumeist die postoperative Behandlung mit Strahlentherapie alleine oder zusammen mit einer Hormontherapie.
Aufgrund der unzureichenden Möglichkeit, Prostatakrebs in aggressive und nicht bzw. weniger aggressive Tumoren zu
klassifizieren, wurde im letzten Jahrzehnt in zahlreichen Studien nach verbesserten Prädiktoren für deren biologisches
Risiko, metastasierten Prostatakrebs zu entwickeln, geforscht. Dabei wurden einzelne Biomarker und Genexpressionssignaturen
getestet. Doch trotz vielfältiger Bemühungen haben die Ergebnisse bislang kaum Anwendung in der klinischen Praxis gefunden.
-
In verschiedenen randomisierten klinischen Studien zu adjuvanter Strahlentherapie gegenüber bloßer Überwachung
bei Patienten mit negativen pathologischen Merkmalen war eine signifikante Abnahme biochemischer Rezidive [1, 2],
lokaler Rezidive [3, 4], und der Metastasierung sowie ein verlängertes Gesamtüberleben für die
Patienten mit adjuvanter Strahlentherapie nachgewiesen worden [3].
Mit konventioneller externer Bestrahlung (60 Gy über sechs Wochen) unmittelbar nach der radikalen Prostatektomie
wurde bei 502 randomisierten Patienten mit pT3-Tumor oder positiven Schnitträndern und hohem Progressionsrisiko
ein signifikant längeres progressionsfreies Überleben und eine bessere lokale Kontrolle erreicht als bei 503
Kontrollpatienten unter „Wait-and-see-Strategie“ [1]. Dieses nach fünf Jahren registrierte
Ergebnis bestätigte sich bei der Analyse eines Follow-up von median 10,6 Jahren bezüglich signifikanter Verbesserung
des biochemischen progressionsfreien Überlebens und der lokalen Kontrolle. Andererseits blieben die Verbesserungen
des klinischen progressionsfreien Überlebens nicht aufrecht erhalten. Aus einer exploratorischen Analyse geht aber
hervor, dass die postoperative Bestrahlung das klinische progressionsfreie Überleben bei Patienten unter 70 Jahren
und bei denen mit positiven Schnitträndern verbessern könnte, aber bei Patienten ab 70 Jahren einen unzuträglichen
Effekt hat [4].
-
Begründet auf der Hypothese, dass sich das biologische Potenzial eines aggressiven Prostatakarzinoms in einer
im Primärtumor entdeckten genomischen Expressionssignatur widerspiegelt, wurde von einem Untersucherteam aus
Mitarbeitern der GenomeDx Biosciences (Vancouver, BC, Kanada), Forschern der Universität Vancouver, der
Universität von Los Angeles, CA und der Mayo Clinic (Rochester, MN) ein genomischer Klassifizierer (GK) mit
22 Markern entwickelt. Hierfür stand von 545 Patienten aus dem Tumorregister der Mayo Clinic (192 Fälle bei
denen ein biochemisches Rezidiv und Fernmetastasen aufgetreten sind und 353 Kontrollen ohne Progression oder
mit biochemischem Rezidiv) RNA zur Verfügung, die für die Analyse erfolgreich auf Microarrays hybridisiert
werden konnte. Für den überwachten Lernprozess wurde ein Satz mit Trainingsdaten von 359 per Zufallsentscheid
ausgewählten Patienten gebildet. Zur Validierung diente der Datensatz der verbliebenen 166 Patienten.
-
Der GK wurde im Trainingsset aus der Analyse von 1,1 Millionen RNA-Features (Positionen auf dem Chip) auf
den Microarrays entwickelt. Nach verschiedenen Selektionsschritten verblieb letztlich ein Satz mit
22 RNA-Biomarkern aus Protein-kodierenden und nicht Protein-kodierenden Regionen des Genoms.
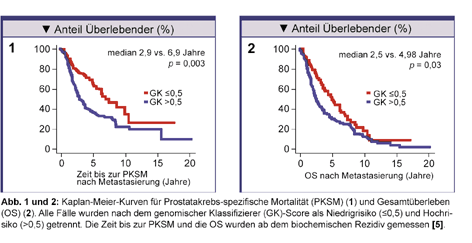
Der GK liefert einen kontinuierlichen Variablenscore zwischen 0 und 1, wobei ein höherer Score auf die
höhere Wahrscheinlichkeit klinischer Metastasierung hinweist. Kaplan-Meier-Kurven der Patientengruppen
(alle Fälle) mit niedrigem und hohem Score des GK zeigen deutliche Unterschiede hinsichtlich des
Prostatakrebs-
Der GK wurde anhand des Validierungssets überprüft. Er wies hinsichtlich seiner prädiktiven Fähigkeit,
frühe klinische Metastasierung zu identifizieren, gegenüber zuvor beschriebenen einzelnen Genen,
Multigen-
Der 22-Feature-GK wird von GenomeDx Biosciences, Vancouver, BC, als Decipher® Prostate Cancer Classifier vermarktet.
Mit dem Test lässt sich gemäß Firmenangaben nach radikaler Prostatektomie anhand der Expression der
22 Biomarker die Wahrscheinlichkeit klinischer Metastasen innerhalb von fünf Jahren und die eines
biochemischen Rezidivs innerhalb von drei Jahren errechnen.
-
Eine der ersten unabhängigen Validierungen des 22-Biomarker-GK erfolgte anhand von Patienten mit negativen
pathologischen Merkmalen nach radikaler Prostatektomie aus dem Tumorregister der Mayo Clinic (nicht identisch
mit den Patienten in der Entwicklungskohorte). Der GK erwies sich in einer multivariaten Analyse als der
überlegene Prädiktor für Metastasierung und Letalität [6].
Zu einem ähnlich positiven Urteil kommen Untersucher aus dem John Hopkins Medical Institute, Baltimore,
MD: Ihre Studienpopulation umfasste 85 Hochrisiko-Patienten, die nach radikaler Pros-tatektomie ein
biochemisches Rezidiv entwickelt hatten. Verglichen mit klinisch-pathologischen Variablen war der
22-Biomarker-GK in der Untersuchungskohorte ein exakterer Prädiktor der metastatischen Progression.
In der multivariaten Analyse war der GK zugleich der alleinige signifikante Prädiktor für Metastasierung [7].
In einer Studienpopulation mit 169 Prostatakrebs-
In einer weiteren retrospektiven Untersuchung wurde der Frage nachgegangen, ob kombinierte Risikomodelle aus
Biomarkern und klinisch-pathologischen Variablen zur Verbesserung der Prädiktion und damit Entscheidungsfindung
nach radikaler Prostatektomie genutzt werden können. Es wurde die prognostische Fähigkeiten von CAPRA-S und
dem 22-Biomarker-GK Decipher® jeweils allein und in Kombination bewertet [9].
Aus einer Kohorte von Prostatakrebs-Patienten mit hohem Rezidivrisiko nach der radikalen Prostatektomie,
die an der Mayo Clinic zwischen 2000 und 2006 behandelt worden waren, konnten in 185 Fällen zugleich Scores
für CAPRA-S und GK bestimmt werden. Siebzehn von ihnen waren am Prostatakrebs verstorben. Für CAPRA-S wurde
ein C-Index von 0,75 und für GK von 0,78 ermittelt. Von 82 der Patienten, die nach dem CAPRA-S-Score 6 mit
hohem Risiko bewertet wurden, hatten 33 auch nach dem GK-Score ein hohes Risiko. Von ihnen starben 17 am
Prostatakrebs. Anhand des GK-Scores wurden die restlichen 49 Patienten mit niedrigem bis intermediärem
Risiko eingestuft. Allerdings verstarben auch von ihnen drei Männer an dem Prostatakrebs. Für Patienten,
die sowohl hohe GK- und hohe CAPRA-S-Scores aufwiesen, betrug die kumulierte Inzidenzrate für
Prostatakrebs-
-
In einer prospektiven, multizentrischen Studie wurde ermittelt, dass der 22-Marker-GK die Therapieentscheidung
bei der Erwägung adjuvanter Strahlentherapie nach radikaler Prostatektomie signifikant beeinflusst und das
 Vertrauen in die Entscheidungsfindung erhöht [10]. Die Studienteilnehmer waren 51 Urologen, deren Aufgabe es
war, Fälle aus den Krankenakten neu zu beurteilen. Bei den vorgelegten Fällen war nach der radikalen
Prostatektomie bei 71% der Patienten ein pT3-Tumor und bei 63% positive Schnittränder gefunden worden. Insgesamt
standen 110 Kasuistiken zum Review zur Verfügung. Für den GK-Test wurden die archivierten Gewebeproben dieser
Patienten herangezogen [10].
Vertrauen in die Entscheidungsfindung erhöht [10]. Die Studienteilnehmer waren 51 Urologen, deren Aufgabe es
war, Fälle aus den Krankenakten neu zu beurteilen. Bei den vorgelegten Fällen war nach der radikalen
Prostatektomie bei 71% der Patienten ein pT3-Tumor und bei 63% positive Schnittränder gefunden worden. Insgesamt
standen 110 Kasuistiken zum Review zur Verfügung. Für den GK-Test wurden die archivierten Gewebeproben dieser
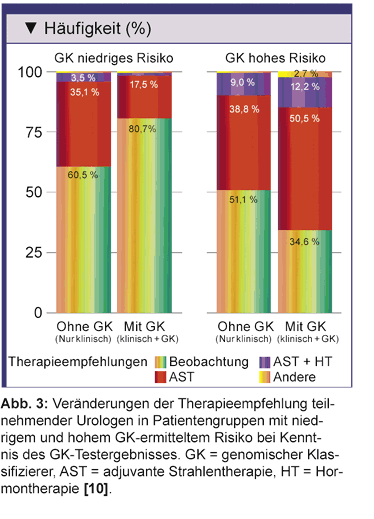
Patienten herangezogen [10].Hatten die Urologen keine Kenntnis des GK-Testergebnisses, wurde für 57% der Patienten anhand der Klinischen Faktoren eine Beobachtung, für 36% eine adjuvante Strahlentherapie und für 7% eine andere Therapie empfohlen. Mit Kenntnis des GK-Testergebnisses wurden 31% andere Therapieentscheidungen getroffen als ohne deren Kenntnis. Davon waren 52% der Wechsel von einer Therapie zur Beobachtung [10]. Bei Fallberichten, die nach GK-Testergebnis als Niedrigrisiko eingestuft wurden, war in 81% der Fälle Beobachtung empfohlen worden. Im Gegensatz dazu war in Fallberichten, die nach GK-Testergebnis als Hochrisiko eingestuft wurden, die Empfehlung zu Beobachtung bei dieser Kenntnis von ursprünglich 51,1% auf 34,6% zurückgegangen (Abb. 3) [10].
Einer weiteren aktuellen Analyse lag die Hypothese zugrunde, dass die Anwendung des genomischen Klassifizierers
zwischen Patienten, die von adjuvanter Strahlentherapie und denen, die von der Salvage-
Anhand der GenomeDx-Gendatenbank wurden die Daten aller Patienten mit einem T3-Prostatakarzinom und/oder
positiven Schnitträndern, die sich an der Thomas Jefferson University, Philadelphia, PA, und der Mayo Clinic
Rochester, MN, zwischen 1990 und 2009 eine Strahlentherapie erhalten hatten, extrahiert [11].
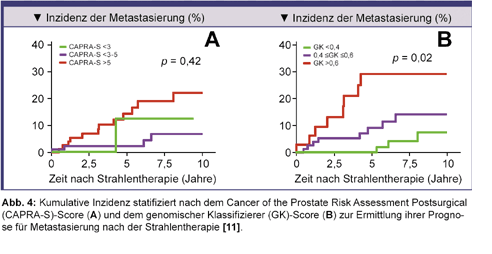
Die kumulativen 5-Jahres-Inzidenzraten bei Patienten mit niedrigem, durchschnittlichem und hohem CAPRA-S-Score
betrugen 13%, 2% bzw. 14% (Abb. 4A). Bei Patienten mit niedrigem, durchschnittlichem und hohem
GK-Score betrugen die kumulativen 5-Jahres-
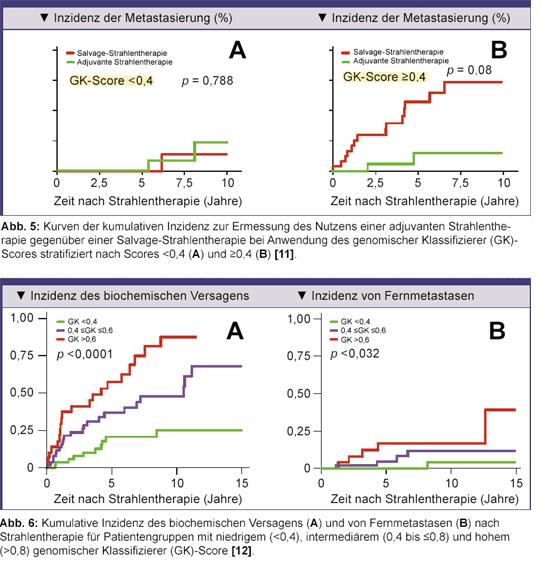
-
Patienten mit einem GK-Score <0,4 hatten sowohl bei adjuvanter als auch bei Salvage-Strahlentherapie die kumulative
5-Jahres-
Im Vergleich zum GK-Score war der CAPRA-S-Score nicht geeignet, Patienten zu identifizieren, die wahrscheinlich von einer adjuvanten Strahlentherapie profitieren würden. Dementsprechend war der C-Index für CAPRA-S 0,66 und 0,83 für GK. Er erhöhte sich in einem kombinierten Modell nur geringfügig auf 0,85 [11].
Überprüft wurde ferner die Hypothese, dass ein genomischer Klassifizierer auch als prädiktives Instrument für biochemisches
Rezidiv und Fernmetastasen bei Prostatakrebs-Patienten dienen kann, die nach der radikalen Prostatektomie eine Strahlentherapie
erhalten. Hierzu wurden Prostatakrebs-Patienten mit pT3-Tumor oder positiven Schnitträndern identifiziert, die sich
am Kimmel Cancer Center, Thomas Jefferson University, Philadelphia, PA, nach radikaler Prostatektomie einer
Strahlentherapie unterzogen hatten. Von 139 Männern lagen Formalin-
-
Von einer Forschergruppe am Massachusetts General Hospital der Harvard University, Boston, MA, wurde ein 32-Gen-
Für die Validierung des Index dienten Gewebeproben einer anderen Gruppe mit 270 Prostatakrebs-
Eine Weichen stellende Therapieentscheidung muss bereits nach jeder positiven Prostatabiopsie bei der Prostatakrebs-
[1] Bolla M, van Poppel H, Collette L et al. 2005. Postoperative radiotherapy after radical prostatectomy: a randomised controlled trial (EORTC trial 22911). Lancet 366:572-678.
[2] Wiegel T, Bottke D, Steiner U et al. 2009. Phase III postoperative adjuvant radiotherapy after radical prostatectomy compared with radical prostatectomy alone in pT3 prostate cancer with postoperative undetectable prostate-specific antigen: ARO 96-02/AUO AP 09/95. J Clin Oncol 27:2924-2930.
[3] Thompson IM, Tangen CM, Paradelo J et al. 2009. Adjuvant radiotherapy for pathological T3N0M0 prostate cancer significantly reduces risk of metastases and improves survival: long-term followup of a randomized clinical trial. J Urol 181:956-962.
[4] Bolla M, van Poppel H, Tombal B et al. 2012. Postoperative radiotherapy after radical prostatectomy for high-risk prostate cancer: long-term results of a randomised controlled trial (EORTC trial 22911). Lancet 380:2018-2027.
[5] Erho N, Crisan A, Vergara IA, et al. 2013. Discovery and validation of a prostate cancer genomic classifier that predicts early metastasis following radical prostatectomy. PLoS ONE 8:e66855.
[6] Karnes RJ, Bergstralh EJ, Davicioni E, et al. 2013. Validation of a genomic classifier that predicts metastasis following radical prostatectomy in an at risk patient population. J Urol 190:2047-2053.
[7] Ross AE, Feng FY, Ghadessi M, et al 2014. A genomic classifier predicting metastatic disease progression in men with biochemical recurrence after prostatectomy. Prostate Cancer Prostatic Dis 17:64-69.
[8] Klein EA, Yousefi K, Haddad ZA, et al. 2015. Genomic classifier improves prediction of metastatic disease within 5 years after surgery in node-negative high-risk prostate cancer patients managed by radical prostatectomy without adjuvant therapy. Eur Urol 67:778-786.
[9] Cooperberg MR, Davicioni E, Crisan A, et al. 2015. Combined value of validated clinical and genomic risk stratification tools for predicting prostate cancer mortality in a high-risk prostatectomy cohort. Eur Urol 67:326-333.
[10] Badani KK, Darby J. Thompson DJ, Brown G, et al. 2015. Effect of a genomic classifier test on clinical practice decisions for patients with high-risk prostate cancer after surgery. BJU Int 115:419-429.
[11] Den RB, Yousefi K, Trabulsi EJ, et al. 2015. Genomic classifier identifies men with adverse pathology after radical prostatectomy who benefit from adjuvant radiation therapy. J Clin Oncol 33:944-951.
[12] Den RB, Feng FY, Showalter TN, et al. 2014. Genomic prostate cancer classifier predicts biochemical failure and metastases in patients after postoperative radiation therapy. Int J Radiation Oncol Biol Phys 89:1038-1046.
[13] Wu C-L, Schroeder BE, Ma X-J, et al. 2013. Development and validation of a 32-gene prognostic index for prostate cancer progression. PNAS 110:6121-6126.
© 2003-2025 pro-anima medizin medien
–
impressum
–
mediadaten
–
konzeption
–
datenschutz

 jfs
jfs